

La familia Filoviridae es una familia de virus del orden Mononegavirales. Su nombre proviene del latín filum (hilo en español), que se refiere a la morfología de los filovirus. Son ARN monocatenario negativo, lo que significa que pertenecen al grupo V de la clasificación Baltimore. Esta familia de virus infecta especialmente a primates. El virus más conocido de este grupo es el Ebolavirus que provoca la enfermedad ébola. Otro filovirus bastante estudiado es el Marburgvirus, mientras que del resto de géneros no se conoce todavía mucho.

En la siguiente lista se podrá observar de forma más detallada el orden taxonómico:
Dentro de la familia Filoviridae aparecen 5 géneros. Su clasificación está designada de esta forma por el análisis filogenético hecho de las secuencias de la ARN polimerasa dependiente de ARN. Los filovirus infectan sobre todo a mamíferos, aunque también hay algunos que infectan a peces.
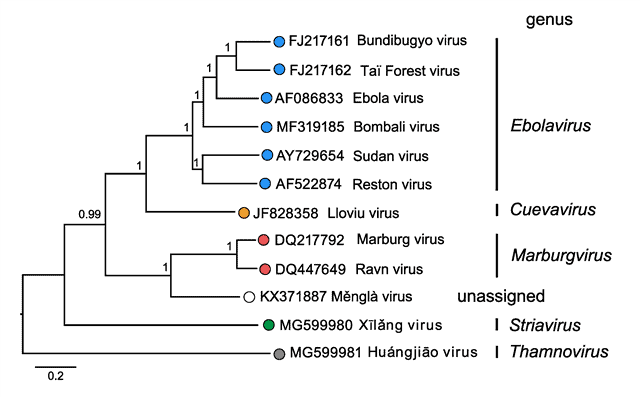
Dentro de la familia Filoviridae aparecen diversas formas, si bien a menudo su forma es filamentosa. Solo se conocen bien las morfologías de los géneros Ebolavirus y Marburgvirus. Además, tienen envuelta. El genoma es de RNA de sentido negativo, linear y no segmentado, y su tamaño oscila entre 15 (Thamnovirus) y 19 kb (Cuevavirus, Ebolavirus, Marburgvirus).
En el caso de los Cuevavirus, parece ser que su hospedador es el murciélago. De hecho, la única especie de este género, Lloviu cuevavirus (LLOV) se descubrió en murciélagos insectívoros muertos de la familia Miniopteridae, concretamente en la especie Miniopterus schreibersii.
Su nombre viene del lugar donde fue descubierto, la Cueva del Lloviu, situada en la provincia de Asturias, España. Aparte del país español, estos virus también aparecieron en especies de murciélagos en Hungría.
Los Ebolavirus tienen 5 especies: Zaire ebolavirus (EBOV), Reston ebolavirus (RESTV), Bundibugyo ebolavirus (BDBV), Sudan ebolavirus (SUDV) y Taï Forest ebolavirus (TAFV), de los cuales 4 (EBOV, BDBV, SUDV y TAFV) provocan infecciones en humanos. No se conocen bien sus hospedadores naturales, si bien se cree que la mayoría tienen a los murciélagos como hospedadores. En el caso de RESTV, también se han encontrado en cerdos. Cada especie es endémica en ciertas zonas del mundo. EBOV y TAFV están en el oeste de África, BDBV y EBOV en el centro de África, BDBV y SUDV en el este de África, mientras que RESTV aparece en el este y sudeste de Asia.
BDBV, EBOV y SUDV son los virus de ébola que causan la enfermedad del ébola en humanos. TAFV también causa enfermedad en humanos, si bien está variante parece ser menos letal que las anteriores. Para su transmisión entre humanos hace falta contacto directo con sangre o fluidos corporales. RESTV infecta a los cerdos, y a primates como Macaca fascicularis, un macaco; a este último le provoca fiebre hemorrágica que podría ser letal.
Su forma es filamentosa aunque también puede ser ramificada, circular en forma de U o de 6. Su longitud varía mucho, pero siempre es de más de 20 μm de largo y tienen un diámetro de entre 96 y 98 nm. Parece ser que el aumento de infectividad de este virus está asociado a una longitud de 805 nm. Su nombre proviene del lugar en el que se registró el primer brote de ébola, el río Ébola de la República Democrática del Congo.
El género Marburgvirus tiene una especie, Marburg marburgvirus, que esta dividida en dos subespecies, Marburg virus (MARV) and Ravn virus (RAVV). Se encuentra en murciélagos pteropódidos como Rousettus aegyptiacus. MARV se distribuye alrededor de África, en las zonas oeste, central, este y sur de este continente, mientras que RAVV está solo en el este de África.
MARV es un patógeno humano con capacidad de ser letal, que provoca la llamada enfermedad de Marburgo. Al igual que el ébola, para su transmisión entre humanos hace falta contacto directo con sangre o fluidos corporales.
Su forma es filamentosa aunque también puede ser ramificada, circular en forma de U o de 6. Su longitud varía mucho, pero siempre es de más de 1 μm de largo y tienen un diámetro de 80 nm. Su nombre provine del lugar donde se registró el primer brote de la enfermedad allá por los años 60', la ciudad alemana de Marburgo.
El género Striavirus tiene una especie, Xilang striavirus (XILV), que se descubrió en peces sapo de la familia Antennariidae. Su nombre proviene del pez sapo que se presupone es su hospedador, Antennarius striatus, en el mar de la China Oriental.
El género Thamnovirus tiene una especie, Huangjiao thamnovirus (HUJV), que se descubrió en lijas, peces de la familia Monacanthidae. Su nombre proviene de la especie en la que se descubrió, Thamnaconus septentrionalis, en el mar de la China Oriental.
La familia Filoviridae tienen RNA como material genético. Expresan entre 6 y 10 proteínas. Su secuencia genómica se expresa desde 3' a 5', y el orden de los genes es 3′-NP-VP35-VP40-(GP)-L-5′. Esta secuencia es muy similar dentro del Filo Negarnaviricota, si bien en los filovirus aparecen genes adicionales. Los genes pueden estar separados por secuencias intergénicas no conservadas o superpuestas. Muchos genes tienen regiones no codificantes relativamente largas.
Los genes de Cuevavirus, Ebolavirus y Marburgvirus están flanqueados por sitios de iniciación y terminación que contienen el pentámero conservado 3'-UAAUU-5'. Los Cuevavirus expresan la ribonucleoproteína del gen VP24 y la ARN polimerasa dependiente de ARN de un ARN mensajero biscistrónico, en vez de usar transcripciones individuales. Los genes GP de los Ebolavirus expresan 3 proteínas diferentes al igual que los Cuevavirus. En cambio, los genes GP de los Marburgvirus codifican una sola proteína, GP1,2.
Los Striavirus no codifican las glicoproteínas de superficie (GP1,2) homólogas a aquellas que si son codificadas por Cuevavirus, Ebolavirus y Marburgvirus.
Los Thamnovirus no codifican los genes VP40 y VP30 que si hacen Cuevavirus, Ebolavirus y Marburgvirus.

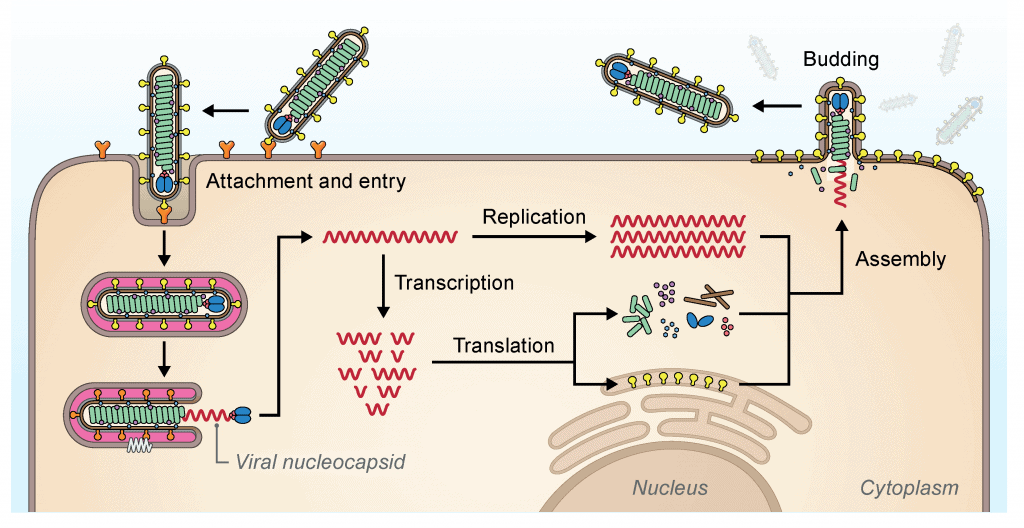
Aquí tienes un esquema sobre la replicación viral de los filovirus. Aquí puedes ver el esquema general de los virus. Verás que hay ciertas diferencias con otras familias de virus. Dentro de los filovirus también hay diferencias. El ciclo de replicación viral que vamos a exponer es el de los Cuevavirus, Ebolavirus y Marburgvirus. No se conocen aún los ciclos de Striavirus y Thamnovirus.
Los virus entran al hospedador por endocitosis. Esto es diferente de la entrada de los coronavirus, por ejemplo. El primer proceso, el de fijación, ocurre cuando estos virus asocian con factores de unión de la membrana plasmática que median la infección por endocitosis. La proteína GP1,2 se encarga de mediar la unión con los factores de unión de la superficie celular. En el caso de los Cuevavirus éstos son las lectinas tipo C, mientras que en Ebolavirus y Marburgvirus pueden ser lectinas tipo C, integrinas, inmunoglobulina de células T y dominios de mucina que contienen la proteína 1 (TIM-1). Después de la unión, se encarga de controlar la bajada de pH dependiente de la fusión en endosomas. En Cuevavirus, Ebolavirus y Marburgvirus es necesaria la unión con el receptor endosomal NCP1.
La transcripción y replicación ocurre en el citoplasma, y en general, estos procesos son similares a los de las familias Paramyxoviridae y Pneumoviridae. La transcripción comienza en el lugar de inicio de la transcripción 3', y la poliadenilación ocurre en un tramo de residuos de uridina (U) dentro del sitio de terminación de la transcripción. Las secuencias no codificantes de la zona terminal 5' favorecen la formación de estructuras similares a las de horquillla en los RNA mensajeros. En el proceso de replicación se sintetiza todo el genoma en sentido positivo (antigenoma) que servirá de molde para la síntesis de proteínas. Además, el genoma original se replicará para formar más material genético que más tarde ser unirá a las proteínas estructurales para formar la nucleocápside.
Durante la infección, se forman una gran cantidad de viriones que se acumular a nivel intracelular y forman cuerpos de inclusión dentro del citoplasma; éstos son los lugares donde ocurren los procesos de transcripción, replicación y formación de la nucleocápside en Ebolavirus y Marburgvirus. Los viriones acabarán saliendo más tarde a la membrana plasmática para adquirir la envuelta; este proceso está controlado por la proteína formada del gen VP40 en Ebolavirus y Marburgvirus.
Albariño, C. G., Wiggleton Guerrero, L., Jenks, H. M., Chakrabarti, A. K., Ksiazek, T. G., Rollin, P. E., & Nichol, S. T. (2017). Insights into Reston virus spillovers and adaption from virus whole genome sequences. PloS one, 12(5), e0178224. https://doi.org/10.1371/journal.pone.0178224
Jens H. Kuhn, Gaya K. Amarasinghe, Christopher F. Basler, Sina Bavari, Alexander Bukreyev, Kartik Chandran, Ian Crozier, Olga Dolnik, John M. Dye, Pierre B. H. Formenty, Anthony Griffiths, Roger Hewson, Gary P. Kobinger, Eric M. Leroy, Elke Mühlberger, Sergey V. Netesov (Нетёсов Сергей Викторович), Gustavo Palacios, Bernadett Pályi, Janusz T. Pawęska, Sophie J. Smither, Ayato Takada (高田礼人), Jonathan S. Towner, Victoria Wahl, and ICTV Report Consortium. 2019, ICTV Virus Taxonomy Profile: Filoviridae, Journal of General Virology, 100, 911–912.
Negredo, A., Palacios, G., Vázquez-Morón, S., González, F., Dopazo, H., Molero, F., Juste, J., Quetglas, J., Savji, N., de la Cruz Martínez, M., Herrera, J. E., Pizarro, M., Hutchison, S. K., Echevarría, J. E., Lipkin, W. I., & Tenorio, A. (2011). Discovery of an ebolavirus-like filovirus in europe. PLoS pathogens, 7(10), e1002304. https://doi.org/10.1371/journal.ppat.1002304
Wang, H., Shi, Y., Song, J., Qi, J., Lu, G., Yan, J., & Gao, G. F. (2016). Ebola Viral Glycoprotein Bound to Its Endosomal Receptor Niemann-Pick C1. Cell, 164(1-2), 258–268. https://doi.org/10.1016/j.cell.2015.12.044

Microbiología© (2023). Todos los derechos reservados