- Microbiología
- Bacterias
- Eucariotas
- Virus
- Blog
Uno de los grandes desafíos actuales que afronta la ciencia es la aparición de bacterias resistentes a los antibióticos. Esto supone la necesidad de descubrir, o bien nuevos antibióticos, o bien otros métodos de eliminación de dichas bacterias. Y, aunque este desafío recae especialmente en la medicina y la microbiología, hay otras ramas científicas que podrían ayudar a la resolución de este problema, como por ejemplo, las ciencias de la computación; concretamente la inteligencia artificial.
Un grupo de investigadores ha conseguido entrenar a una red neuronal para que tenga la capacidad de predecir moléculas con capacidad antibacteriana. Para ello, han realizado predicciones en diversas bibliotecas químicas, y se ha descubierto un compuesto con la capacidad antibacteriana deseada, la halicina.
Índice de contenidos
Este hallazgo llega en el mejor momento, ya que desde hace décadas no se descubre ningún nuevo antibiótico; solo aparecen nuevas generaciones de antibióticos ya existentes. Esto, junto con la aparición cada vez más común de bacterias resistentes a los antibióticos, hace que la necesidad de encontrar nuevos antibióticos sea urgente. Se han están probando otras alternativas, como renovar el uso bacteriófagos en terapia antimicrobiana. Pero otra opción es encontrar nuevos antibióticos con ayuda de la computación.
No es nuevo el intento usar la informática para encontrar nuevos antibióticos. Ya en el pasado se han desarrollado modelos para predicciones de las propiedades de ciertas moléculas. No obstante, la falta de precisión de estos modelos hacia que estos intentos fueran en vano. Ahora bien, los nuevos avances de los algoritmos de los modelos neuronales hacen que aparezca una oportunidad para descubrir nuevos medicamentos. Ahora, la red neuronal es capaz de predecir las propiedades de una molécula de manera precisa, si bien todavía hay diferencias entre los enfoques analítico y experimental. Entonces, la mejor solución sería unir ambos enfoques.
Esto es lo que hicieron este grupo de investigadores. Primero, entrenaron a un modelo de red neuronal para predecir la inhibición del crecimiento de Escherichia coli usando una colección de 2.335 moléculas. Después, aplicaron este modelo a un conjunto de librerías químicas que englobaban más 107 millones de moléculas, para identificar compuestos con potencial actividad contra E. coli. Por último, tras clasificar a dichos compuestos moleculares en función de la puntuación otorgada por el modelo de predicción, se seleccionaron una lista de candidatos viables.
Mediante este método, identificaron un compuesto de una librería química llamada 'Drug Repository Hub', concretamente el inhibidor SU3327 de la kinasa c-jun N-teminal, renombrado como halicina. Este compuesto, diferente a nivel estructural a los antibióticos convencionales, se mostró como un potente inhibidor del crecimiento de E. coli.
Del mismo modo, la halicina muestra actividad bactericida frente a un amplio espectro de bacterias infecciosas, que incluyen a Mycobacterium tuberculosis (bacteria de la tuberculosis) y Enterobacterias resistentes a carbapenem. Además, también ha demostrado su efectividad contra Clostridioides difficile y Acinetobacter baumannii pan-resistentes en modelos murinos (modelo de ratones para investigación). Esta propiedad antibacteriana es debido a que interrumpe el flujo de protones a través de la membrana celular. Además, en los experimentos mostró una baja toxicidad y dificultad a la aparición de resistencias.
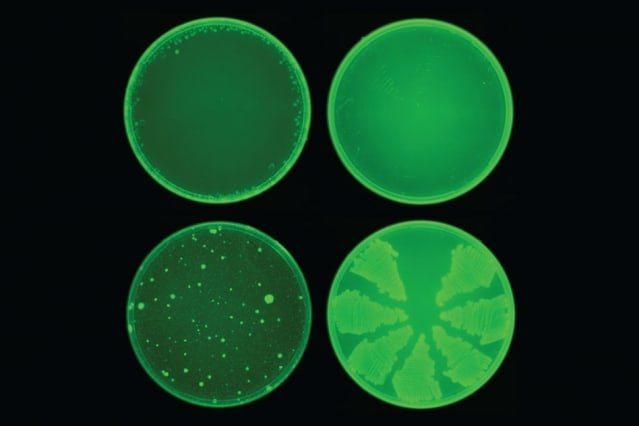
Además de la halicina, se han identificado ocho componentes antibacterianos que son estructuralmente diferentes de los antibióticos conocidos. De esas moléculas, dos mostraron actividad antibiótica de amplio espectro, y podrían tener la capacidad de vencer a cepas de E. coli resistentes a antibióticos.
Los modelos neuronales no son perfectos; hay que diseñar el entrenamiento de manera precisa para nuestro objetivo, se deben elegir bien las librerías químicas, y se debe tener claro cuales son las prioridades en las posibles predicciones. Aun con sus limitaciones, el uso de modelos de redes neuronales tiene el potencial de encontrar nuevos compuestos antibióticos con menos recursos, abaratando el coste de las investigaciones. Este no deja de ser un estudio preliminar, pero nos indica un posible camino hacia el descubrimiento de nuevos antibióticos, y la resolución del problema de las bacterias resistentes a éstos.
Jonathan M. Stokes et al. A deep learning approach to antibiotic discovery. Cell, vol. 180, n.º 4, págs. 688-702 (2020). DOI: https://doi.org/10.1016/j.cell.2020.01.021