

La taxonomía es la ciencia (dentro de la biología) que se encarga de ordenar a los seres vivos usando elementos clasificatorios llamados taxones. Vamos a ver como están organizados, además de la forma científica de nombrar a las especies (hay diferencias en función de si es animal, vegetal, bacteria, etc).
El orden taxonómico actual, de mayor a menor, es el que aparece en la siguiente imagen.
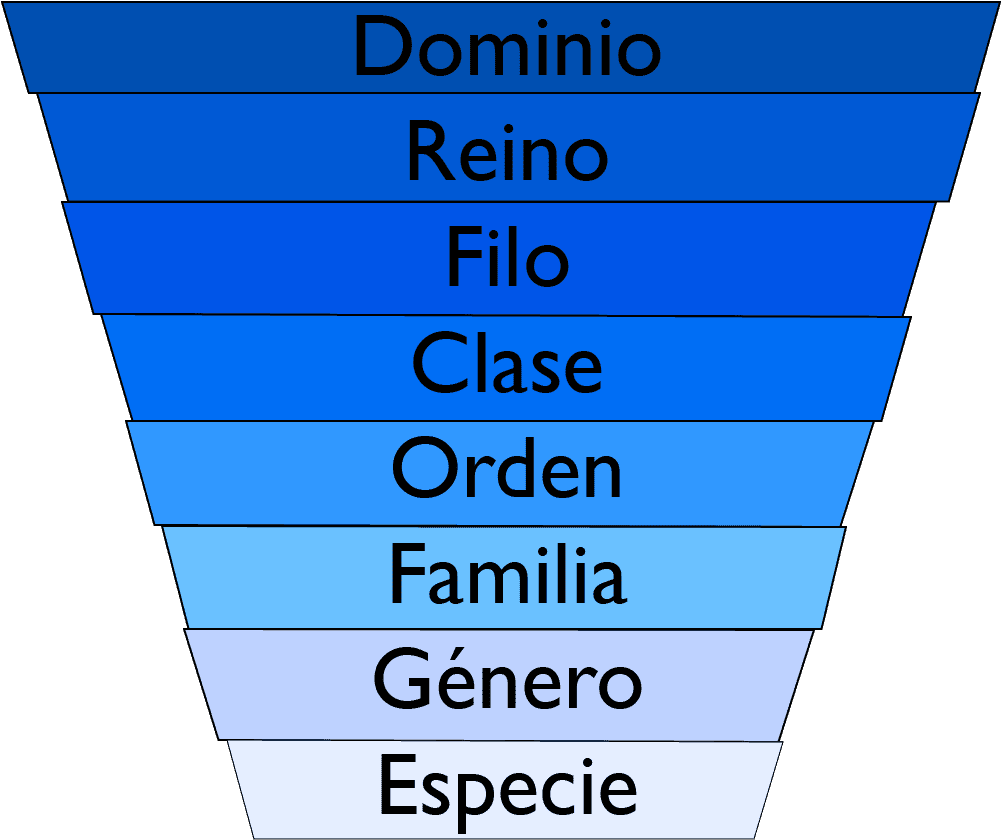
Cada grupo que aparece aquí (dominio, reino, etc) es un taxón. Son grupos de clasificación para los seres vivos que se organizan en función de sus semejanzas físicas y genéticas. Vamos a hablar de las categorías actuales. Y sí, la taxonomía es una ciencia en constante cambio. Sin ir más lejos, hasta hace unos años, tanto Bacteria como Archaea estaban dentro del antiguo reino Monera. No obstante, ahora mismo la organización taxonómica es la siguiente:
Mas adelante veremos varios ejemplos para entender mejor esta clasificación.
Dentro de cada taxón pueden haber categorías intermedias (subreinos en un reino, agrupación de familias formando una superfamilia, etc). Estas categorías intermedias son opcionales, y se usan para que la clasificación este mejor organizada y sea más fácil encontrar el taxón de interés en el caso de que haya muchos elementos dentro de un grupo. Esta sería la clasificación taxonómica usando dichas categorias intermedias:
Pueden variar en función de si son animales, plantas o bacterias; de hecho, hay códigos de nomenclatura cada uno de éstos. Actualmente, tenemos estos códigos de nomenclatura:
Ahora bien, también hay clasificaciones en función de su forma de vida, de su metabolismo, etc. Por ejemplo, se pueden clasificar a los seres vivos en función de su fuente de energía, poder reductor y carbono.
Se encarga de establecer normas para un correcto nombramiento de los animales, dando así una forma común para todos los zoólogos del mundo. Los nombres se dan en latín. Hay terminaciones específicas para el Reino Animalia que afectan a los taxones inferiores a familia (incluyendo familia).
Los taxones superiores no tienen terminaciones específicas. Así, los filos Porifera o Cnidaria, las clases Calcarea o Hydrozoa, o los órdenes Clathrinida o Lemnomedusae no tienen terminaciones comunes. Pueden aparecer terminaciones comunes dentro de un taxón, pero no todos los filos, clases y órdenes tienen las mismas terminaciones.
Este código da una forma común de nombrar a los taxones de los reino Plantae, Protista y Fungi. En el siguiente recuadro se pueden ver las terminaciones que aparecen en estos reinos. Como se puede ver, hay terminaciones comunes para determinados taxones.
| Categoría taxonómica | Plantae | Protista | Fungi |
| Filo | -phyta | -mycota | |
| Subfilo | -phytina | -mycotina | |
| Clase | -opsida | -phyceae | -mycetes |
| Subclase | -idae | -phycidae | -mycetidae |
| Superorden | -anae | ||
| Orden | -ales | ||
| Suborden | -ineae | ||
| Infraorden | -aria | ||
| Superfamilia | -acea | ||
| Familia | -aceae | ||
| Subfamilia | -oideae | ||
| Tribu | -eae, -ae | ||
| Subtribu | |||
Las terminaciones del Reino Bacteria son:
Este método de nomenclatura se basa en dos nombres, llevando el primero la letra inicial en mayúscula, mientras que el segundo está en minúsculas (Escherichia coli, Canis lupus). Es el científico sueco Carl von Linneo quien comienza a generalizar el uso de esta forma de nomenclatura.
Cada especie tiene un nombre compuesto por dos partes:
También pueden escribirse con 3 palabras; en ese caso estaríamos hablando de nomemclatura trinomial. Se usan para nombrar una subespecie dentro de una especie. Así, por ejemplo, podemos diferenciar al lobo ibérico (Canis lupus signatus) del lobo común (Canis lupus lupus).
Los nombres se escriben en latín y no van acompañados de artículos. Siempre que se escriban deben resaltarse, así que normalmente aparecen en cursiva. Esto es sencillo en documentos web; en caso de escritura a mano se puede subrayar (Canis lupus). Si usamos el nombre común y el científico de una especie a la vez el nombre científico aparece entre paréntesis. Ejemplo: El lince ibérico (Lynx Pardinus) es una especie endémica de la península ibérica.
Además, se debe escribir el nombre completo la primera vez que hablamos de dicha especie; en las siguientes veces siguientes que nos refiramos a dicha especie podemos usar su forma abreviada. Así, ahora sería correcto escribir E. coli o C. lupus, ya que hemos escrito su nombre completo anteriormente.
Si estudias biología en la universidad casi que, con total seguridad, vas a tener que aprender usar esta clasificación. Ese examen es conocido como visu (palabra del latín, cuyo significado es "visto directamente"). Este examen suele hacerse en zoología y botánica. En ese examen te pedirán, como máximo, los 8 principales taxones de clasificación, si bien normalmente pedirán los taxones descendentes a partir del filo (6 taxones). No obstante, en alguno de los ejemplos que vamos a ver usaremos también algún taxón intermedio (y así verás que pueden ser bastante útiles). Verás que se suele escribir en forma de lista, y los taxones van en orden descendente. No obstante, el género no se escribe solo; va acompañado del nombre de la especie en cuestión, usando la nomenclatura binaria. Vamos a explicar las características de los taxones que se usen.
Vamos a ver su uso con las siguientes especies: El elefante asiático (Elephas maximus), el búho real (Bubo bubo), el camarón gris (Crangon crangon), el bígaro común (Littorina littorea), La carabela portuguesa (Physalia physalis) y la lavanda común (Lavandula angustifolia).
Las primeras 5 especies entran dentro del reino Animalia, mientras que la lavanda está en el reino Plantae. Ahora viene la lista de dichas especies:
Elephas maximus
Bubo bubo
Crangon crangon
Littorina littorea
Physalia phisalis
Lavandula angustifolia
La pandemia de SARS-CoV-2 ha provocado muchos cambios a nivel mundial. Los confinamientos, toques de queda, y en resumen, restricciones a la libre circulación de personas se han dado en casi todos los países del mundo. Y la razón por la que se han dado es que parecía ser que esta enfermedad podía ser transmisible por personas asintomáticas, o sea, personas con el virus en su interior que no desarrollan síntomas, pero que parecía ser que tenían capacidad de contagiar.
Pero, ¿y si no es así? ¿Y si los asintomáticos no contagian? Esto es lo que ha aparecido en un estudio en el que se llevó a cabo un cribado a casi toda la población de Wuhan en el periodo post-confinamiento.
El estudio de Cao S. et al., (2020) describe el programa de cribado (screening en inglés) llevado a cabo en Wuhan en el mes de mayo de este año. En este estudio se hizo el test al 90% de la población, casi 10 millones de personas.
El screening no mostró nuevos casos de COVID-19 entre los participantes que no hubieran pasado la enfermedad previamente, y sí que mostró 300 nuevos positivos asintomáticos. De estos 300 casos asintomáticos, se rastrearon sus contactos cercanos, 1174 personas. Todas dieron negativo en test de COVID-19. De los 34424 casos de personas recuperadas de COVID-19, 107 dieron positivo de nuevo.
Se hicieron cultivos virales para todos los casos asintomáticos y positivos de personas recuperadas. Las pruebas indicaron que no habían "virus viables" en ninguno de estos casos.
Esto significaría un cambio en el paradigma de la lucha contra COVID-19. Las medidas llevadas a cabo para limitar la expansión de COVID-19 han sido especialmente para detener la transmisión de esta enfermedad por personas asintomáticas. Problablemente irán apareciendo más estudios sobre el tema, y seguiremos teniendo más información. Seguiremos informando.
Cao, S., Gan, Y., Wang, C. et al. Post-lockdown SARS-CoV-2 nucleic acid screening in nearly ten million residents of Wuhan, China. Nat Commun 11, 5917 (2020). https://doi.org/10.1038/s41467-020-19802-w
La clasificación del Comité Internacional de Taxonomía de Virus (siglas ICTV en inglés) utiliza una forma de clasificar muy parecida a la que se hace con los seres vivos. Así, los virus se clasifican por orden, familia, subfamilia, género y especie.
Los virus se clasifican en 4 dominios, 9 reinos y 16 filos, además de 1 orden, y diversas familias y géneros que no están englobados en ningún dominio.
El dominio Duplodnaviria engloba virus ADN de doble cadena que codifican la cápside proteica HK97.
El dominio Monodnaviria engloba a los virus ADN de una cadena.
El dominio Riboviria engloba a los virus que codifican polimerasas dependientes de ARN (virus ARN).
El dominio Varidnaviria engloba diversos virus ADN de doble cadena.
Las terminaciones son las siguientes:
Esta es la taxonomía mediante la clasificación ICTV actual:
La familia Filoviridae es una familia de virus del orden Mononegavirales. Su nombre proviene del latín filum (hilo en español), que se refiere a la morfología de los filovirus. Son ARN monocatenario negativo, lo que significa que pertenecen al grupo V de la clasificación Baltimore. Esta familia de virus infecta especialmente a primates. El virus más conocido de este grupo es el Ebolavirus que provoca la enfermedad ébola. Otro filovirus bastante estudiado es el Marburgvirus, mientras que del resto de géneros no se conoce todavía mucho.

En la siguiente lista se podrá observar de forma más detallada el orden taxonómico:
Dentro de la familia Filoviridae aparecen 5 géneros. Su clasificación está designada de esta forma por el análisis filogenético hecho de las secuencias de la ARN polimerasa dependiente de ARN. Los filovirus infectan sobre todo a mamíferos, aunque también hay algunos que infectan a peces.
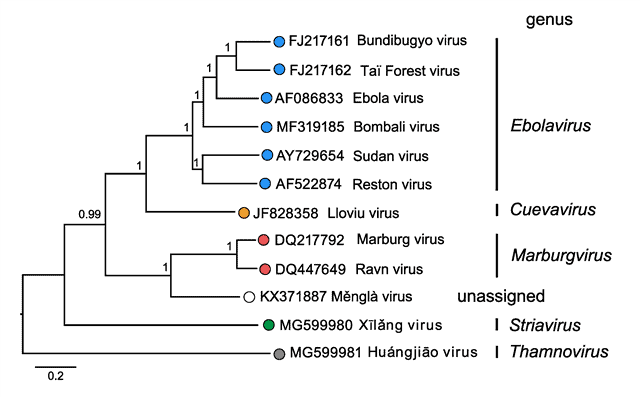
Dentro de la familia Filoviridae aparecen diversas formas, si bien a menudo su forma es filamentosa. Solo se conocen bien las morfologías de los géneros Ebolavirus y Marburgvirus. Además, tienen envuelta. El genoma es de RNA de sentido negativo, linear y no segmentado, y su tamaño oscila entre 15 (Thamnovirus) y 19 kb (Cuevavirus, Ebolavirus, Marburgvirus).
En el caso de los Cuevavirus, parece ser que su hospedador es el murciélago. De hecho, la única especie de este género, Lloviu cuevavirus (LLOV) se descubrió en murciélagos insectívoros muertos de la familia Miniopteridae, concretamente en la especie Miniopterus schreibersii.
Su nombre viene del lugar donde fue descubierto, la Cueva del Lloviu, situada en la provincia de Asturias, España. Aparte del país español, estos virus también aparecieron en especies de murciélagos en Hungría.
Los Ebolavirus tienen 5 especies: Zaire ebolavirus (EBOV), Reston ebolavirus (RESTV), Bundibugyo ebolavirus (BDBV), Sudan ebolavirus (SUDV) y Taï Forest ebolavirus (TAFV), de los cuales 4 (EBOV, BDBV, SUDV y TAFV) provocan infecciones en humanos. No se conocen bien sus hospedadores naturales, si bien se cree que la mayoría tienen a los murciélagos como hospedadores. En el caso de RESTV, también se han encontrado en cerdos. Cada especie es endémica en ciertas zonas del mundo. EBOV y TAFV están en el oeste de África, BDBV y EBOV en el centro de África, BDBV y SUDV en el este de África, mientras que RESTV aparece en el este y sudeste de Asia.
BDBV, EBOV y SUDV son los virus de ébola que causan la enfermedad del ébola en humanos. TAFV también causa enfermedad en humanos, si bien está variante parece ser menos letal que las anteriores. Para su transmisión entre humanos hace falta contacto directo con sangre o fluidos corporales. RESTV infecta a los cerdos, y a primates como Macaca fascicularis, un macaco; a este último le provoca fiebre hemorrágica que podría ser letal.
Su forma es filamentosa aunque también puede ser ramificada, circular en forma de U o de 6. Su longitud varía mucho, pero siempre es de más de 20 μm de largo y tienen un diámetro de entre 96 y 98 nm. Parece ser que el aumento de infectividad de este virus está asociado a una longitud de 805 nm. Su nombre proviene del lugar en el que se registró el primer brote de ébola, el río Ébola de la República Democrática del Congo.
El género Marburgvirus tiene una especie, Marburg marburgvirus, que esta dividida en dos subespecies, Marburg virus (MARV) and Ravn virus (RAVV). Se encuentra en murciélagos pteropódidos como Rousettus aegyptiacus. MARV se distribuye alrededor de África, en las zonas oeste, central, este y sur de este continente, mientras que RAVV está solo en el este de África.
MARV es un patógeno humano con capacidad de ser letal, que provoca la llamada enfermedad de Marburgo. Al igual que el ébola, para su transmisión entre humanos hace falta contacto directo con sangre o fluidos corporales.
Su forma es filamentosa aunque también puede ser ramificada, circular en forma de U o de 6. Su longitud varía mucho, pero siempre es de más de 1 μm de largo y tienen un diámetro de 80 nm. Su nombre provine del lugar donde se registró el primer brote de la enfermedad allá por los años 60', la ciudad alemana de Marburgo.
El género Striavirus tiene una especie, Xilang striavirus (XILV), que se descubrió en peces sapo de la familia Antennariidae. Su nombre proviene del pez sapo que se presupone es su hospedador, Antennarius striatus, en el mar de la China Oriental.
El género Thamnovirus tiene una especie, Huangjiao thamnovirus (HUJV), que se descubrió en lijas, peces de la familia Monacanthidae. Su nombre proviene de la especie en la que se descubrió, Thamnaconus septentrionalis, en el mar de la China Oriental.
La familia Filoviridae tienen RNA como material genético. Expresan entre 6 y 10 proteínas. Su secuencia genómica se expresa desde 3' a 5', y el orden de los genes es 3′-NP-VP35-VP40-(GP)-L-5′. Esta secuencia es muy similar dentro del Filo Negarnaviricota, si bien en los filovirus aparecen genes adicionales. Los genes pueden estar separados por secuencias intergénicas no conservadas o superpuestas. Muchos genes tienen regiones no codificantes relativamente largas.
Los genes de Cuevavirus, Ebolavirus y Marburgvirus están flanqueados por sitios de iniciación y terminación que contienen el pentámero conservado 3'-UAAUU-5'. Los Cuevavirus expresan la ribonucleoproteína del gen VP24 y la ARN polimerasa dependiente de ARN de un ARN mensajero biscistrónico, en vez de usar transcripciones individuales. Los genes GP de los Ebolavirus expresan 3 proteínas diferentes al igual que los Cuevavirus. En cambio, los genes GP de los Marburgvirus codifican una sola proteína, GP1,2.
Los Striavirus no codifican las glicoproteínas de superficie (GP1,2) homólogas a aquellas que si son codificadas por Cuevavirus, Ebolavirus y Marburgvirus.
Los Thamnovirus no codifican los genes VP40 y VP30 que si hacen Cuevavirus, Ebolavirus y Marburgvirus.

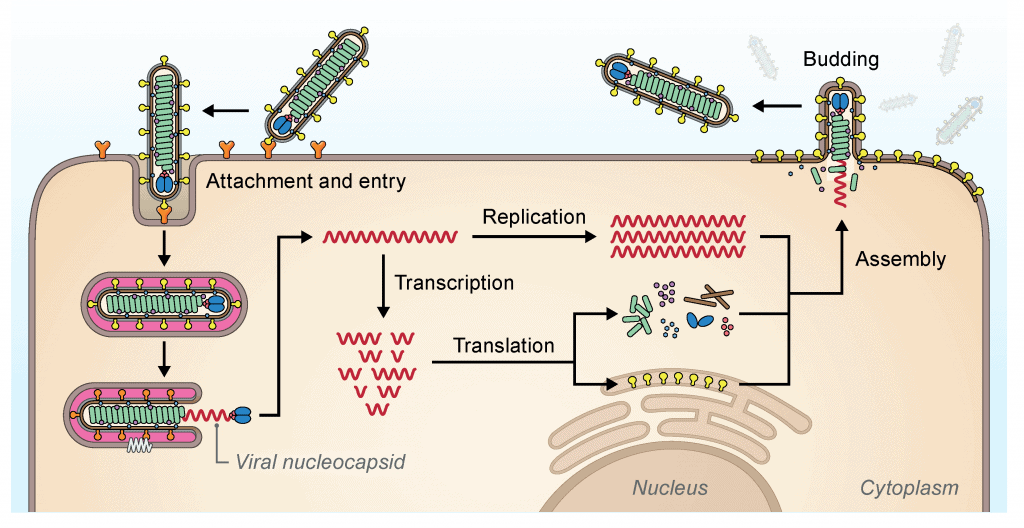
Aquí tienes un esquema sobre la replicación viral de los filovirus. Aquí puedes ver el esquema general de los virus. Verás que hay ciertas diferencias con otras familias de virus. Dentro de los filovirus también hay diferencias. El ciclo de replicación viral que vamos a exponer es el de los Cuevavirus, Ebolavirus y Marburgvirus. No se conocen aún los ciclos de Striavirus y Thamnovirus.
Los virus entran al hospedador por endocitosis. Esto es diferente de la entrada de los coronavirus, por ejemplo. El primer proceso, el de fijación, ocurre cuando estos virus asocian con factores de unión de la membrana plasmática que median la infección por endocitosis. La proteína GP1,2 se encarga de mediar la unión con los factores de unión de la superficie celular. En el caso de los Cuevavirus éstos son las lectinas tipo C, mientras que en Ebolavirus y Marburgvirus pueden ser lectinas tipo C, integrinas, inmunoglobulina de células T y dominios de mucina que contienen la proteína 1 (TIM-1). Después de la unión, se encarga de controlar la bajada de pH dependiente de la fusión en endosomas. En Cuevavirus, Ebolavirus y Marburgvirus es necesaria la unión con el receptor endosomal NCP1.
La transcripción y replicación ocurre en el citoplasma, y en general, estos procesos son similares a los de las familias Paramyxoviridae y Pneumoviridae. La transcripción comienza en el lugar de inicio de la transcripción 3', y la poliadenilación ocurre en un tramo de residuos de uridina (U) dentro del sitio de terminación de la transcripción. Las secuencias no codificantes de la zona terminal 5' favorecen la formación de estructuras similares a las de horquillla en los RNA mensajeros. En el proceso de replicación se sintetiza todo el genoma en sentido positivo (antigenoma) que servirá de molde para la síntesis de proteínas. Además, el genoma original se replicará para formar más material genético que más tarde ser unirá a las proteínas estructurales para formar la nucleocápside.
Durante la infección, se forman una gran cantidad de viriones que se acumular a nivel intracelular y forman cuerpos de inclusión dentro del citoplasma; éstos son los lugares donde ocurren los procesos de transcripción, replicación y formación de la nucleocápside en Ebolavirus y Marburgvirus. Los viriones acabarán saliendo más tarde a la membrana plasmática para adquirir la envuelta; este proceso está controlado por la proteína formada del gen VP40 en Ebolavirus y Marburgvirus.
Albariño, C. G., Wiggleton Guerrero, L., Jenks, H. M., Chakrabarti, A. K., Ksiazek, T. G., Rollin, P. E., & Nichol, S. T. (2017). Insights into Reston virus spillovers and adaption from virus whole genome sequences. PloS one, 12(5), e0178224. https://doi.org/10.1371/journal.pone.0178224
Jens H. Kuhn, Gaya K. Amarasinghe, Christopher F. Basler, Sina Bavari, Alexander Bukreyev, Kartik Chandran, Ian Crozier, Olga Dolnik, John M. Dye, Pierre B. H. Formenty, Anthony Griffiths, Roger Hewson, Gary P. Kobinger, Eric M. Leroy, Elke Mühlberger, Sergey V. Netesov (Нетёсов Сергей Викторович), Gustavo Palacios, Bernadett Pályi, Janusz T. Pawęska, Sophie J. Smither, Ayato Takada (高田礼人), Jonathan S. Towner, Victoria Wahl, and ICTV Report Consortium. 2019, ICTV Virus Taxonomy Profile: Filoviridae, Journal of General Virology, 100, 911–912.
Negredo, A., Palacios, G., Vázquez-Morón, S., González, F., Dopazo, H., Molero, F., Juste, J., Quetglas, J., Savji, N., de la Cruz Martínez, M., Herrera, J. E., Pizarro, M., Hutchison, S. K., Echevarría, J. E., Lipkin, W. I., & Tenorio, A. (2011). Discovery of an ebolavirus-like filovirus in europe. PLoS pathogens, 7(10), e1002304. https://doi.org/10.1371/journal.ppat.1002304
Wang, H., Shi, Y., Song, J., Qi, J., Lu, G., Yan, J., & Gao, G. F. (2016). Ebola Viral Glycoprotein Bound to Its Endosomal Receptor Niemann-Pick C1. Cell, 164(1-2), 258–268. https://doi.org/10.1016/j.cell.2015.12.044
Los principales síntomas del COVID-19 son fiebre, tos y dificultad para respirar. No obstante, también pueden aparecer otros síntomas del tracto respiratorio o gastrointestinales entre otros, si bien la proporción es menor.
Para llegar al diagnóstico de COVID-19 se hace un aspirado nasal o traqueal. Esto también se puede usar para descartar la presencia de gripe. Para confirmar la presencia de SARS-CoV-2 se hace la llamada reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR). Si la persona da positivo para la enfermedad pueden pasar dos cosas: cuarentena en casa (síntomas leves) o en el hospital (síntomas más fuertes o mayor riesgo de mortalidad). En caso de que se quede en el hospital se deberán seguir haciendo otras pruebas para observar la evolución de la enfermedad en el paciente. Se pueden hacer tanto pruebas bioquímicas como captar imágenes de los pulmones.
Después de esto, lo normal sería hacer el típico examen médico, con un recuento sanguíneo completo (RSC) y un panel metabólico completo (PMC), en especial chequeando posibles marcadores asociados a un aumento de la mortalidad en COVID-19.
Cuando hay una infección, lo normal es tener un recuento de glóbulos blancos alto, pero en el caso de COVID-19 hay linfopenia (recuento bajo de glóbulos blancos). El panel metabólico nos puede ayudar a detectar posibles fallos multiorgánicos (¿recuerdas los que podía ocurrir en casos graves?). En caso de fallo en la perfusión del hígado, tenemos como señales el aumento de la bilirrubina y enzimas hepáticas como AST (aspartato aminotransferasa) y ALT (alanina aminotransferasa). Si hay fallo en los riñones aparecerán un aumento de BUN (nitrógeno ureico en sangre) y creatinina. Si hay fallo en el corazón aparecerá un aumento de las troponinas y de la creatinkinasa MB (CK-MB).
También se pueden buscar marcadores de la inflamación que, si bien son generales, pueden ayudar a hacernos una idea de la situación del paciente. Así, aparecerían altas concentraciones de CRP (proteína C reactiva), de la tasa de sedimentación eritrocítica (ESR), interleucina 6 (IL-6), lactato deshidrogenasa (LDH), dímero-d y ferritina. Si un paciente tiene neumonía viral aumenta el riesgo de coger una infección bacteriana. En caso de que, además de la infección por COVID-19, haya una infección causada por bacterias aparecerá una concentración alta de PCT (procalcitocina).
Además de las pruebas bioquímicas, también se pueden sacar imágenes. Muy a grosso modo: en todas las imágenes que pueden aparecer, si la zona de los pulmones está oscura, es que todo va bien. Si aparecen cosas claras, hay problemas.
Rayos X. No son los más útiles, pero se pueden ver cosas. La parte inferior de los pulmones tiene lo que se llama 'opacidad en vidrio esmerilado' (GGO), un área nebulosa de aumento de la atenuación del pulmón con marcas bronquiales y vasculares preservadas; estas zonas deberían aparecer oscuras, pero no es el caso. Esto es indicador de que los pulmones no están bien, pero hay más cosas que pueden aparecer en los pulmones, y que no alcanzamos a ver con rayos X. Hacen falta otras tecnologías.

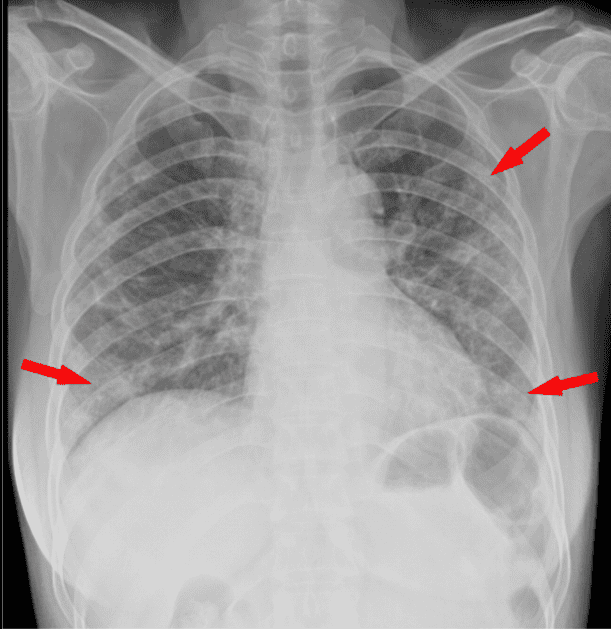
Escáner por tomografía computerizada (TC). Probablemente es la tecnología que puede darnos una perspectiva más completa a día de hoy. Aquí también se pueden ver:
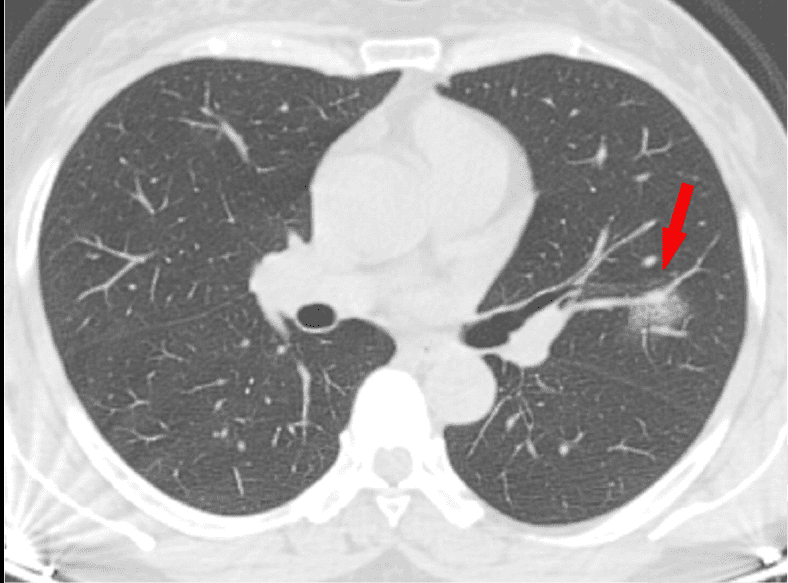
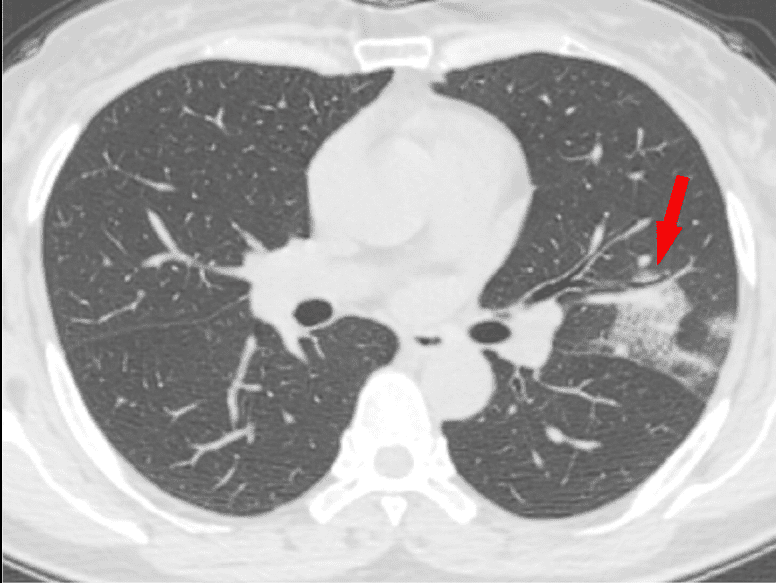

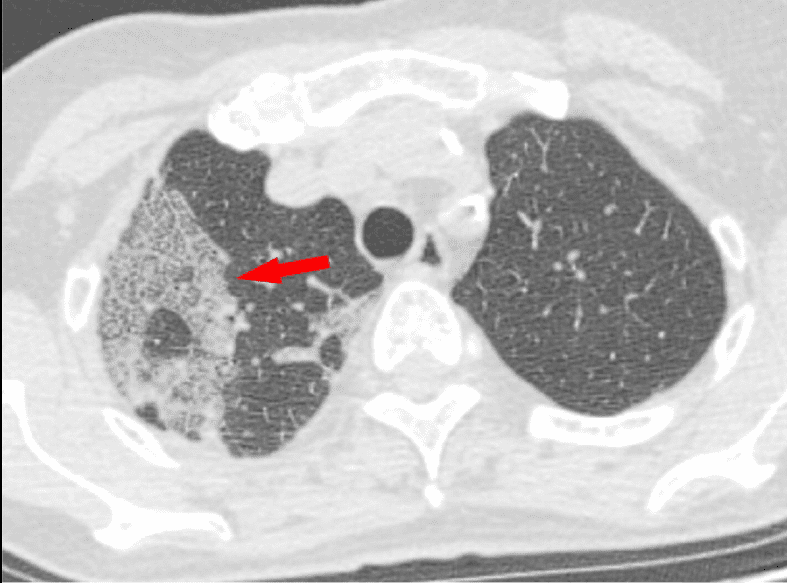


Ecografía. Los ultrasonidos son útiles para buscar espesamiento pleural, aumento de las líneas B (indicador de edema pulmonar), consolidaciones con broncograma aéreo, y flujo sanguíneo deficiente.
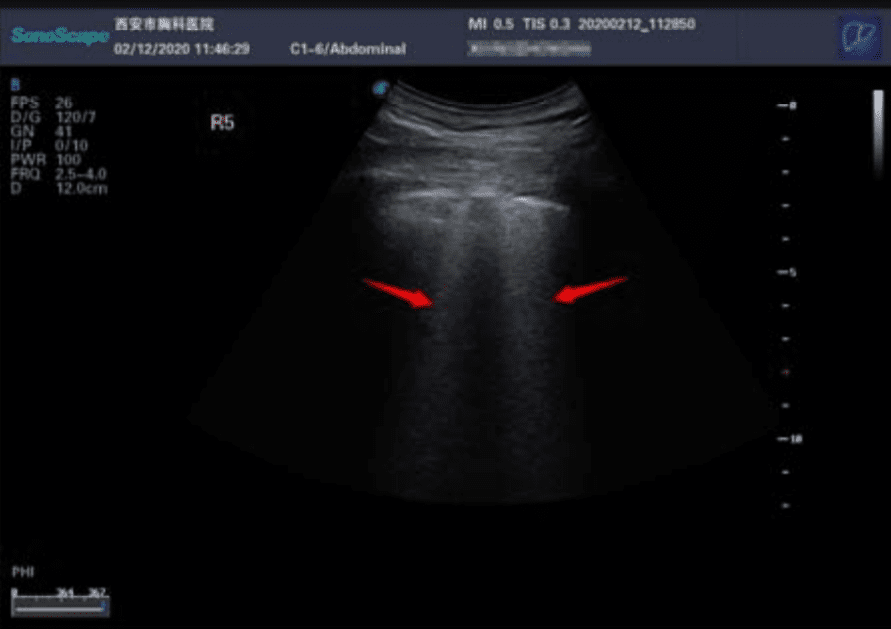
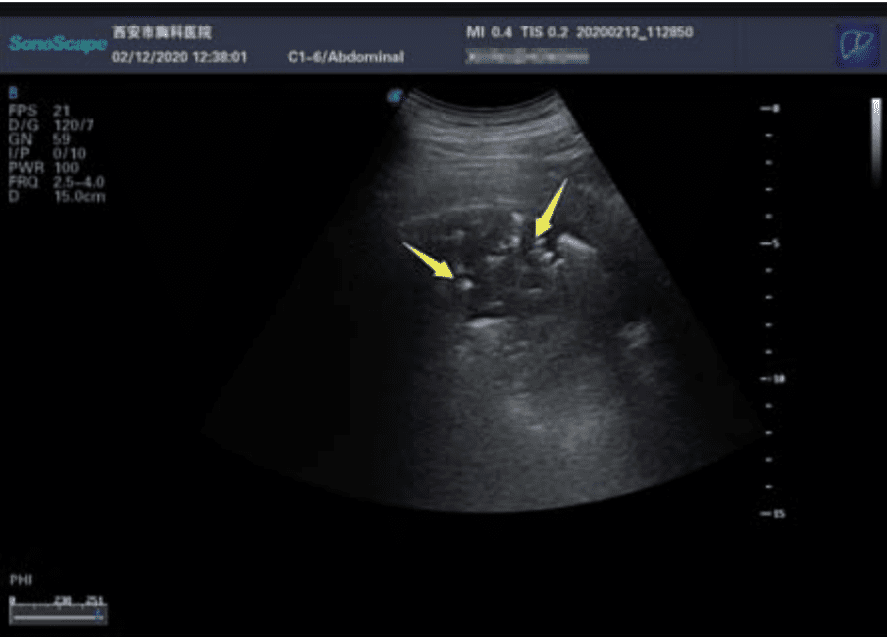
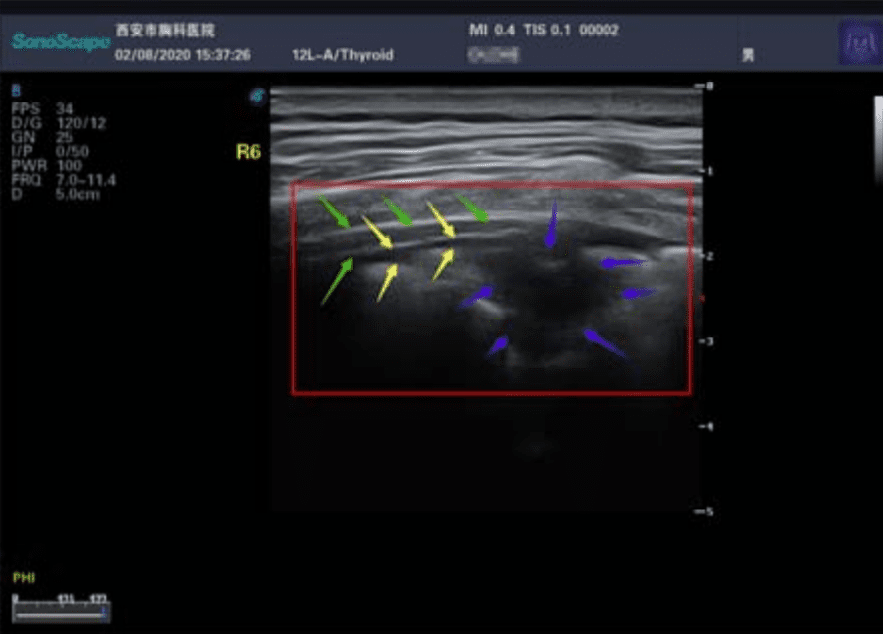
Huang Y., Sihan Wang, Yue Liu, Yaohui Zhang, Chuyun Zheng, Yu Zheng, Chaoyang Zhang, Weili Min, Ming Yu, Mingjun Hu. A preliminary study on the ultrasonic manifestations of peripulmonary lesions of non-critical novel coronavirus pneumonia (COVID-19). SSRN (2020) doi: 10.21203/rs.2.24369/v1
Ye, Z., Zhang, Y., Wang, Y. et al. Chest CT manifestations of new coronavirus disease 2019 (COVID-19): a pictorial review. Eur Radiol (2020). https://doi.org/10.1007/s00330-020-06801-0
Zi Yue Zu, Meng Di Jiang, Peng Peng Xu, Wen Chen, Qian Qian Ni, Guang Ming Lu, and Long Jiang Zhang. Coronavirus Disease 2019 (COVID-19): A Perspective from China Radiology 0 0:0 (2020) https://doi.org/10.1148/radiol.2020200490
Es el tiempo en el que tardan en aparecer los síntomas en dos personas con el virus, tanto en la primera persona que infecta como en la segunda persona infectada. En otras palabras, es el tiempo en el que tarda un agente infeccioso en propagarse. Cuanto más corto sea ese número, más peligroso es el agente infeccioso.
El intervalo en serie varía según los estudios que mires, pero los números oscilan entre 5 y 7.5 días en COVID-19, mientras que en la gripe común es de 2.5 días. El intervalo en serie de COVID-19 es alto, si, pero el verdadero problema reside en que, como ya se demostró hace meses, una persona infectada puede trasmitir la enfermedad aún sin mostrar síntomas. Así que la velocidad de propagación es más alta de la que los números dicen.
El llamado R0 o ritmo reproductivo básico es el número promedio de casos nuevos que genera un caso dado a lo largo de un período infeccioso. En otras palabras, el R0 indica el número de personas que puede contagiar una persona infectada.
Cuanto más grande sea ese número, hay más probabilidades de que dicha infección pueda formar un brote epidémico. Este número es distinto en cada virus. El R0 medio de COVID-19 es 2.5, si bien factores como el calor y la humedad pueden reducir ese número.
Entonces, hagamos una pequeña simulación. En el peor de los casos, 1 persona infecta a 3 personas. Esas 3 personas infectan a otras (ya son 9); estas 9 infectan a otras 3 (ya son 27); estas 27 a otras 3 (81), de 81 pasamos a 243, 729... El crecimiento es exponencial.
El R0 de la gripe común es 1.3. Si hacemos la misma simulación, vemos que el aumento es lineal, de 1 en 1 la mayoría de casos. De ahí podemos concluir que la tasa de transmisión de COVID-19 es mucho más alta que la de la gripe común.
Actualización: según el CDC, el R0 de COVID-19 sin ninguna medida de contención es de 5.7. Si el crecimiento con un R0 de 3 era exagerado, más de 5 es demasiado. Por ello hay que tomar medidas.
Por supuesto. Estas son las medidas a tomar en para virar la curva:
Estas y otras medidas ayudarán a prevenir COVID-19. Aquí tienes un artículo con más detalles.

Pero como se ha comentado antes, el clima puede jugar a nuestro favor. Al igual que en el caso de la gripe común, COVID-19 ha demostrado reducir su R0 con el aumento de la temperatura y la humedad relativa. Así, el clima veraniego y la temporada de lluvias puede ayudar a reducir la viabilidad de SARS-CoV-2.

Wang, Jingyuan and Tang, Ke and Feng, Kai and Lv, Weifeng.
High Temperature and High Humidity Reduce the Transmission of COVID-19 (March 9, 2020). Available at SSRN: http://dx.doi.org/10.2139/ssrn...
Respuesta técnica: el jabón disuelve la bicapa lipídica de los virus, lo que provoca que éstos se inactiven. No podemos decir que los virus "mueren" ya que, realmente, no son seres vivos. Vamos a explicar todo este proceso, pero antes de nada hay que conocer mejor a los virus.
Los virus están formados por proteínas, lípidos y material genético, que puede ser ADN o ARN. En el caso del coronavirus SARS-CoV-2 tiene ARN. El ARN es el lugar donde se almacena la información para la formación de nuevos virus. Las proteínas tienen diversas funciones: se encargan de la unión al hospedador, ayudan al proceso de replicación, y hay otras con funciones estructurales. Los lípidos tienen función estructural, 'protegen' al virus. Las unidades estructurales del virus se unen por enlaces no covalentes que no necesitan de energía para su formación, ya que la interacción entre estas unidades estructurales es energéticamente más estable que si las unidades estuvieran libres. Así, la estructura se regulariza y es muy eficiente, muy díficil de romper.
Los coronavirus tienen un tamaño que oscila entre 50-200 nanómetros; así que se puede decir que son nanopartículas. Las nanopartículas tienen complejas interacciones con las superficies en las que estén, y esto ocurre con los virus también. Cuando un virus invade un hospedador (como una célula humana), el virus usa la maquinaria celular del huésped para su proceso de replicación; se replica el material genético y las proteínas. No obstante, los lípidos los obtienen de la célula huésped, y así, el ARN y las proteínas se autoensamblan con los lípidos. Por cierto, no se forma solo un virus, se forman muchísimas copias, y todos estos nuevos virus saturan la célula, que acaba muriendo. La célula estalla y libera todos estos nuevos que seguirán este mismo proceso para seguir replicándose (Esto es un resumen del ciclo de replicación vírico; aquí hay una explicación más detallada).

En los pulmones, algunos de estos virus terminan en las vías respiratorias, y en las membranas mucosas que los envuelven. Y así, cuando toses (y aún más cuando estornudas) se expulsan gotas respiratorias al exterior que pueden llegar a recorrer hasta 10 metros. En el caso de los coronavirus, parece ser que pueden llegar a los 2 metros. Por todo esto, ¡debes cubrirte cuando tosas o estornudes! Estas gotas suelen secarse rápido cuando acaban en cualquier superficie. Pero hay un problema: los virus siguen activos.
Principio químico: las moléculas similares entre si parece ser que interactúan entre ellas de forma más intensa que aquellas que son diferentes entre sí. Por ejemplo, la madera, la tela o la piel interactúan y mucho con los virus. En el caso contrario tenemos al acero, la porcelana y algunos plásticos como el teflón. La estructura de la superficie también importa; así, superficies ásperas pueden separar al virus. La estructura vírica está unida mediante una combinación de enlaces de hidrógeno e interacciones hidrofílicas. Así, las superficies compuestas por fibras o la madera (por ejemplo) pueden formar muchos enlaces de hidrógeno con el virus. Por contra, el acero, el teflón o la porcelana no forman muchos enlaces de hidrógeno; el virus no se une fuertemente a estas superficies. Entonces, los virus tenderán a ir hacia aquellas superficies donde puedan formar más enlaces.
SARS-CoV-2 parece que permanece activo en superficies favorables durante horas, o incluso durante 1 día. La humedad, la luz del sol (radiación UV) y el calor hacen que el virus sea menos estable.
La piel es una superficie ideal para el virus, ya que es 'orgánica', y las proteínas y lípidos de las células muertas de la superficie de la piel interactúan con el virus a través de enlaces de hidrógeno e interacciones hidrofílicas. Así, cuando tocas una superficie de acero con partículas virales en él, éstos se pegarán a tu piel; se quedarán en tus manos.
De momento no son peligrosos, no estás infectado. Ahora bien, si te tocas la cara con las manos, el virus podría transmitirse de las manos a la cara. Entonces, los virus están cerca de las vías respiratorias y de las membranas mucosas (nariz, ojos, boca). El virus ahora si que puede entrar a tu cuerpo, y si, estás infectado, a menos que el sistema inmune mate al virus. Además, puedes transmitir el virus con las manos, la boca, las gotas respiratorias. Un apretón de manos, besos... todo esto puede ayudar a la propagación del virus.
Piensa en lo normal que es llevarse las manos a la cara. Hasta que no te laves las manos, corres riesgo de infectarte. Veamos varios elementos con los que puedes lavarte las manos.
Solo agua. Podría funcionar. PERO... el agua 'solo' es capaz de romper los enlaces de hidrógeno formados entre el virus y la piel. Hay más enlaces, así que es difícil que los virus se despeguen y se vayan. Hace falta algo más.
Agua con jabón. Esto ya es distinto. El jabón está formado por moléculas anfipáticas: esto significa que tienen un extremo hidrófilo, soluble al agua, y otro hidrófobo, que rechaza el agua y se une a los lípidos. Este extremo hidrófobo se asemeja a los lípidos de la membrana de los virus. Las moléculas de jabón 'compiten' con los lípidos de la membrana vírica. Además, también compiten con otros enlaces no covalentes que ayudan a mantener la estructura del virus. El jabón es efectivo 'disolviendo' las uniones que mantienen a las estructuras víricas.
El jabón también compite con las interacciones entre el virus y la superficie de la piel con su extremo hidrófilo. Así, la combinación de agua y jabón nos quita a los virus de la piel, y los virus se van. La piel es bastante áspera y con arrugas; por ello, hay que tomarse el tiempo necesario para frotar y remojar las manos por todas partes, ya que los virus pueden estar en cualquier zona de la piel. Estas propiedades, unidas a las del agua, hacen que esta sea la mejor opción.
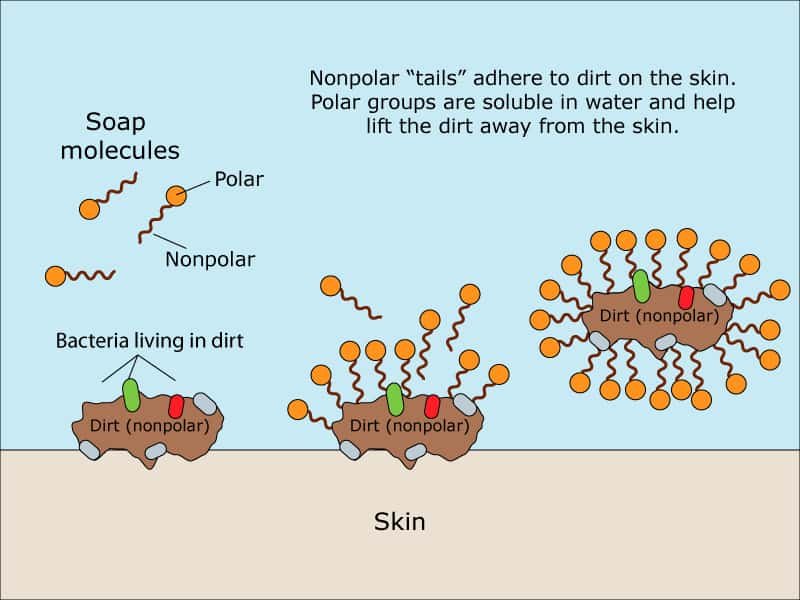
Productos desinfectantes basados en alcohol. Los productos basados en alcohol, que suelen incluir propiedades desinfectantes y antibacterianas, contienen un gran procentaje de alcohol (entre 60-80% de etanol), a veces con algo de isopropanol, además de agua y un poco de jabón. El etanol y otros alcoholes no solo forman facílmente enlaces de hidrógeno con los virus; además, como disolvente, son más lipofílicos que el agua. De ahí que el alcohol disuelva la membrana lipídica y pueda romper ciertas interacciones de los virus. Casi todos los productos antibacterianos contienen alcohol y algo de jabón, lo que ayuda a matar a los virus. Pero algunos incluyen agentes antibacterianos 'activos' como el triclosán, que no afectan en nada a los virus.
No obstante, hace falta una alta concentración de alcohol (+60%) para conseguir una rápida disolución del virus. Por ejemplo, el vodka o el whisky (~40%) no disolverían el virus tan rápido. En general, el alcohol no es tan efectivo como el jabón para este hacer; además, el alcohol es más corrosivo para la piel que el jabón.
En resumen, el lavado de manos, aún siendo algo muy rutinario y sencillo, es una acción de gran importancia para prevenir virus (también bacterias). Lavarse las manos solo con agua puede ayudar, y los geles hidroalcohólicos son efectivos contra los virus, pero hace falta un porcentaje de alcohol alto. Por cierto, si el gel hidroalcohólico tiene alguna propiedad antibacteriana, ésta no es efectiva contra los virus. Si no estás en condición de usar jabón se pueden usar, son válidos; pero si puedes, lo más efectivo es lavarse las manos con agua y jabón.

Fuente: Palli Thordarson, profesor de química en la Universidad de Nueva Gales del Sur, en su cuenta personal de twitter.
La familia Hepeviridae pertenece al grupo IV de la clasificación Baltimore. Esto significa que son virus ARN monocatenarios positivos (ssRNA+); su material genético es ARN lineal con un tamaño de entre 6.6-7.2 kb. Son virus esféricos desnudos, carecen de envoltura, y su diamétro oscila entre los 27-34 nanómetros. En el microscopio electrónico se pueden observar picos y hendiduras de los viriones.
La cápside está formada por capsómeros formados por homodímeros de una sola proteína de cápside. Cada proteina de cápside contiene tres dominios lineares que forman distintas estrcuturas en la cápside. El dominio S forma la cápside en si misma, el dominio P1 forma protuberancias triples y el dominio P2 forma dobles picos, además de tener epítopes neutralizantes. Además, parece ser que cada dominio cuenta con lugares de unión a polisacáridos que podrían interactuar con los receptores celulares.

Son virus animales; afectan a mamíferos, aves y peces. Tienen dos géneros y cinco especies. Esta su taxonomía:
El Orthohepevirus suele ser más conocido como Hepevirus. Cabe destacar que el Orthohepevirus A es el causante de la hepatitis E.
Los virus que infectan a mamíferos son los Orthohepevirus A, Orthohepevirus C y Orthohepevirus D, mientras que Orthohepevirus B infecta a aves. Piscihepevirus A infecta a peces (truchas).
Como ya se ha comentado, la familia Hepevirus infecta a mamíferos, aves y peces. Su transmisión es entérica, lo que significa que se puede transmitir de persona a persona por via fecal-oral, por bebidas o alimentos contaminados, siendo la más común la contaminación del agua por heces fecales. También puede transmitirse por el consumo de carne cruda o poco hecha, y en menor probabilidad, a través de transfusiones de sangre.
Orthohepevirus A y Orthohepevirus B se asocian a infecciones agudas en el hígado. La hepatitis E crónica se ha convertido en un problema clínico de gran importancia en individuos inmunodeprimidos, en especial en los pacientes a los que les van a trasplantar órganos. Del resto de especies no se conoce bien la el modo de transmisión.
La hepatitis E es una enfermedad hepática (del hígado) que se produce por la infección de Orthohepevirus A, o dicho de forma más sencilla, el virus de la hepatitis E. Se conocen cuatro genotipos: el 1 y el 2 se han encontrado solo en humanos, mientras que el 3 y el 4 aparecen en algunos animales, aunque pueden infectar a humanos.
Son virus desnudos, aunque se ha observado que pueden obtener una cubierta (por la membrana celular del huésped) durante el proceso de ensamble y salida al exterior, lo que podría ayudar al virus a no ser reconocido por los anticuerpos.
Como el resto de virus de la familia Hepeviridae, se transmite por vía fecal-oral. La mayoría de casos de infección han sido provocados por el consumo de agua con contaminación fecal. En menor medida, han aparecido casos provocados por el consumo de productos cárnicos poco cocinados de animales infectados, por transfusiones de sangre y por transmisión maternofetal.
La aparición de hepatitis E es frecuente es países de ingresos medios y bajos, donde la calidad del agua no está bien controlada y es más fácil que aparezcan contaminaciones. En estas zonas aparece el genotipo 1 con mayor frecuencia que el genotipo 2. Suelen aparecer en brotes, con la contaminación del agua para beber, afectando a miles de personas. También pueden aparecer de manera esporádica.
En los países con mejor calidad de vida, no suelen haber muchos casos, y la mayoría son casos esporádicos provocados por el genotipo 3. El genotipo 3 aparece en productos cárnicos poco cocinados; los animales que tienen esta infección, aunque a ellos no les provoque enfermedad, si que lo hace en humanos.
Los síntomas pueden durar entre 1 y 6 semanas. La fase inicial comienza con fiebre leve, disminución del apetito, náuseas y vómitos, que dura pocos días. En algunos casos puede aparecer dolor abdominal, prurito (picor en la piel), erupciones cutáneas o dolores articulares. Más tarde, aparecen la ictericia (coloración amarillenta de la piel y la esclerótica de los ojos) además de orina oscura y heces claras, y hepatomegalia (ligero aumento del tamaño del hígado), con dolor a la palpación.
Estos síntomas no difieren de los otros tipos de hepatitis, por lo que es necesario un diagnóstico correcto para determinar si se trata de hepatitis E.
A veces, esta enfermedad puede degenerar en hepatitis fulminante, que puede llegar a ser mortal. Esta evolución de la enfermedad es más frecuente durante el embarazo, pudiendo ser letal para la madre y el feto.
La hepatitis E se suele detectar mediante la detección en la sangre de anticuerpos IgM específicos contra ese virus. Esta forma de diagnosticar es común en los lugares donde esta infección es frecuente.
Otra forma de diagnóstico, más avanzada, es mediante la reacción en cadena de la polimerasa con retrotranscriptasa (RT-PCR), que detecta el ARN del virus en la sangre o las heces. Esta prueba se hace en laboratorios especializados, y es necesaria en los lugares donde la infección no es frecuente y en casos de pacientes con infección crónica.
A día de hoy no hay tratamientos específicos contra la hepatitis E aguda. No obstante, se ha usado ribavirina para tratar a pacientes inmunodeprimidos con hepatitis E crónica, además de interferón en algunas ocasiones.
La enfermedad suele remitir de forma espontánea, así que la mayoría de veces no es necesario hospitalizar al paciente, e incluso se debe evitar el uso de ciertos fármacos como el paracetamol o antieméticos. Ahora bien, sí que se debe hospitalizar en caso de hepatitis fulminante.
A nivel individual se puede prevenir la hepatitis E mediante la correcta higiene y evitando consumir agua o productos cárnicos en caso de no estar seguros de si están contaminados o no.
A nivel colectivo se puede prevenir mediante el correcto funcionamiento de los sistemas de desechos de heces y de los sistemas de suministros de agua.
Hepeviridae - Hepeviridae - Positive-sense RNA Viruses - International Committee on Taxonomy of Viruses (ICTV). (2020) https://talk.ictvonline.org/ictv-reports/ictv_online_report/positive-sense-rna-viruses/w/hepeviridae
Hepatitis E (WHO) (2020) https://www.who.int/es/news-room/fact-sheets/detail/hepatitis-e
La clasificación del Comité Internacional de Taxonomía de Virus (siglas ICTV en inglés) utiliza una forma de clasificar muy parecida a la que se hace con los seres vivos. Así, los virus se clasifican por orden, familia, subfamilia, género y especie.
Solo existe el reino Riboviria, grupo taxonómico que agrupa a los virus ARN y los viroides. De la clasificación de Baltimore están representados los grupos III, IV y V. La primera parte de su nombre ribo proviene de ácido ribonucleico (ARN). Es el primer reino de virus propuesto. En el futuro puede añadirse los virus de ADN pero se sigue investigando.
Actualización: Los virus se clasifican en 4 dominios, 9 reinos y 16 filos. Para ver la nueva clasificación, haz clic aquí.
Las terminaciones son las siguientes:
Esta es la taxonomía mediante la clasificación ICTV actual:
Para ver la nueva clasificación, puedes hacerlo en nuestra nueva página de la clasificación ICTV actualizada.
El Sistema de Clasificación de Baltimore fue propuesto por David Baltimore. Está basada en el tipo de ácido nucleico de los virus (ADN o ARN) y su modo de expresión génica; en otras palabras, se clasifica según su material genético.
Si has visto el ciclo de los virus, habrás observado que el proceso de replicación de los virus difería en función de su grupo. En nuestra página de replicación de los virus puedes ver las diferencias de cada grupo.
En este artículo verás expresiones a las abreviaturas en inglés de ADN y ARN: DNA y RNA. Si eres estudiante de biología, microbiología, o algo similar esto no te pillará de sorpresa. No obstante, puede que haya gente que le siga sonando más natural la abreviatura española...conforme se avanza en los estudios, estas cosas cambian 😅
Según la Clasificación Baltimore, los virus se distribuyen en siete grupos:
Son virus DNA de doble cadena. La replicación del DNA del virus se realiza por medio de las DNA-polimerasas dependiente de DNA del huésped o codificadas por el virus. Son los virus más comunes, y los más diversos. Esta es su taxonomía:
Son virus DNA de una cadena. Al igual que el grupo I, la replicación del DNA del virus se realiza por medio de las DNA-polimerasas dependiente de DNA. La cadena de DNA puede ser diferente en los virus, así que dentro de este grupo los virus se pueden clasificar en:
Esta es su taxonomía, formada por familias aún no asignadas:
Son virus RNA de doble cadena. Los virus de este grupo se replican en el citoplasma y no dependen de las polimerasas de las células huésped, a diferencia de los virus DNA, ya que incluyen las enzimas necesarias en el virión. Esta es su taxonomía:
Son virus RNA de una cadena. Los virus de este grupo se replican en el citoplasma; a diferencia de los virus DNA, no son tan dependientes del huésped, ni usan DNA intermedio para replicarse. Su cadena de RNA tiene polaridad positiva lo que significa que son idénticos al mRNA viral, así que pueden ser traducidos inmediatamente por el hospedador. Esta es su taxonomía:
Son virus RNA de una cadena. Los virus de este grupo no usan DNA intermedio para replicarse. El RNA viral es negativo, lo que significa que es complementario del mRNA, así que debe convertirse en RNA positivo por una RNA polimerasa antes de la traducción. Puesto que es necesario el proceso de transformación del RNA a positivo, el RNA 'original' del virus no es en si mismo infeccioso. Aquí se incluyen también a los virus RNA monocatenarios ambisentido (sentido postivo y negativo: virus ssRNA(+/-)). Esta es su taxonomía:
Son virus RNA de una cadena. Lo que tiene de especial este grupo de virus es que se replica mediante un proceso llamado 'transcripción inversa': se forma DNA a partir del RNA original por medio de una enzima llamada transcriptasa inversa. En este grupo hay se usa un intermediario de DNA aunque sea un virus RNA, a diferencia de los virus RNA vistos anteriormente. Esta es su taxonomía:
Son virus DNA de doble cadena. Este grupo de virus replica el material genético mediante la 'transcripción inversa': se forma RNA a partir del DNA original; este RNA vuelve a convertirse en DNA por medio de la transcriptasa inversa. En este grupo hay se usa un intermediario de RNA aunque sea un virus DNA, a diferencia de los virus DNA vistos anteriormente. Esta es su taxonomía:

Microbiología© (2023). Todos los derechos reservados