Familia Coronaviridae
La familia Coronaviridae son virus ARN monocatenario positivo (virus +ssRNA), por lo que pertenecen al grupo IV de la clasificación Baltimore. El tamaño de su genoma oscila entre 27 y 31 kb. Está familia está formada por los coronavirus y la especie Microhyla letovirus 1, englobada en otra subfamilia. Los coronavirus infectan de manera frecuente a los humanos; en especial, las cepas de coronavirus 229E, NL63, OC43 y HKU1, que provocan síntomas de un resfriado común.
Durante el año 2020 (sobre todo) se ha estado hablando mucho de los coronavirus debido a la reciente infección por el nuevo coronavirus (SARS-CoV-2) que provoca la enfermedad llamada COVID-19; si bien otras especies de coronavirus ya han causado infecciones graves tales como el síndrome respiratorio agudo grave (SARS) o el síndrome respiratorio de oriente medio (MERS). Se organizan dentro del orden Nidovirales, en la familia Coronaviridae. Se caracterizan por su proteína S o de espiga, que forma picos fuera de la envuelta, y les da la apariencia de corona; de ahí su nombre.
Todos los coronavirus tienen similaridades en la organización y expresión de su genoma. Aparecen 16 proteínas no estructurales (nsp1-nsp16) codificadas por un marco de lectura abierto (ORF) 1a/b en el extremo terminal 5'. También aparecen las proteínas estructurales S o de espiga, E o de envoltura, M o de membrana y N o nucleocápside, codificadas por otros ORFs en el extremo terminal 3'.
En la siguiente lista se podrá observar de forma más detallada el orden taxonómico:
Índice de contenidos
Taxonomía de la familia Coronaviridae
- Orden Nidovirales
- Suborden Cornidoviridae
- Familia Coronaviridae
- Subfamilia Letovirinae
- Género Alphaletovirus
- Subfamilia Orthocoronavirinae
- Género Alphacoronavirus
- Género Betacoronavirus
- Género Deltacoronavirus
- Género Gammacoronavirus
- Subfamilia Letovirinae
- Familia Coronaviridae
- Suborden Cornidoviridae
En el género Betacoronavirus aparecen 4 lineajes (A, B, C, and D). A diferencia del resto, el lineaje A de los beta-CoV también codifican también una proteína llamada hemaglutinin esterasa (HE), que tiene una funcionalidad parecida a la de la proteína S.
Subfamilia Letovirinae (Alphaletovirus)
No confundir con la subfamilia Lentivirinae, anteriormente ubicada dentro de la familia Retroviridae. Actualmente esa subfamilia está en desuso, pero sí que existe el género Lentivirus del cual destaca la especie del VIH.
La especie Microhyla letovirus 1 (MLeV) se ha descubierto en Microhyla fissipes, un tipo de rana (anfibio anuro). Esta especie se engloba dentro de la subfamilia Letovirinae, en el género Alphaletovirus, y es la única representante de estos taxones. Como primera opción se propusó el nombre de Microhyla alphaletovirus 1, pero al final sufrió una pequeña variación. El nombre de esta especie (letovirus) proviene del personaje mítico de la cultura griega 'Latona' (Leto en inglés). La historia cuenta que Latona convirtió a algunos campesinos en ranas después de que éstos no dejaran beber del agua de un lago. Curioso, cuanto menos.
El estudio de su secuencia respaldó la hipótesis de que esta nueva especie podía ser hermana de los coronavirus (subfamilia Orthocoronavirinae), con algunas diferencias frente a éstos, pero dentro de la familia Coronaviridae. Por ejemplo, en la región pp1a, aparecieron dominios típicos de los coronavirus, como los dominios carboxilterminales nsp4, Mpro, nsp7, nsp8, nsp9 y nsp10. En la región que se encarga de las estructuras proteicas, se detectó un dominio aminoterminal de los coronavirus en la proteína N de MLeV.
Subfamilia Orthocoronavirinae
Origen de los coronavirus
Los coronavirus pertenecientes a la subfamilia Orthocoronaviridae (a partir de ahora los llamaremos coronavirus a secas) infectan a un amplio rango de animales, además de a los humanos. No obstante, el origen principal de los coronavirus parecen ser los murciélagos, de ahí que se piense que éstos son el mayor reservorio de alfacoronavirus y betacoronavirus. Para que alguno de estos coronavirus llegue a infectar a los humanos, debe pasar primero por procesos de recombinación que les haga adaptarse a otro animal, que hace de intermediario entre los murciélagos y nosotros. Los animales domésticos, debido a su cercanía, pueden ser intermediarios, y, de hecho, son susceptibles de infectarse con coronavirus, como lo muestran las infecciones en cerdos por PEDV y SADS-CoV.
En el caso de SARS-CoV, el coronavirus causante del SARS, se trata de un virus que empezó en los murciélagos. Algún virus de murciélago relacionados con el SARS (SARSr-CoV) sufrió un proceso de recombinación, por el cual pudo infectar a las civetas (el intermediario) y llegar al hospedador final, los humanos, en 2002. He ahí la clave, la recombinación, la capacidad de adaptación a nuevos huéspedes, lo que hace que estos coronavirus puedan infectar a un amplio rango de especies.
En el caso de MERS-CoV, el coronavirus causante del MERS, es otro virus que comienza en los murciélagos. En este caso, el intermediario fue el dromedario. Parece ser que este virus se adaptó para infectar a los dromedarios hace al menos 30 años. Más tarde, en 2012, adquirieron la capacidad de infectar a los humanos.
En el caso de H-CoV-229E y HCoV-NL63, coronavirus causantes del resfriado común, parece ser que su origen está en murciélagos africanos y los camélidos serían el intermediario de H-CoV-229E. Aún no se conoce el intermediario de HCoV-NL63. En el caso de HCoV-OC43 y HKU1, otros coronavirus causantes del resfriado común, parece que tienen su origen en los roedores.
El caso de SARS-CoV-2 todavía tiene interrogantes. El origen, como en la mayoría de casos, parece ser el murciélago. Pero para que llegue a humanos, hace falta un intermediario; sino, este sería el primer caso en el que un coronavirus pasa directamente de murciélagos a humanos. Se ha hablado mucho de los pangolines, pero por el momento están descartados. De momento la historia más "creible" es la de la sopa de murciélago. No obstante, hace falta más investigación para acabar descubriendo los orígenes de SARS-CoV-2.

Estructura genómica
Los coronavirus tienen entre 100-160 nm de diámetro. Son esféricos y tienen envuelta. Como ya hemos comentado son virus ARN de una cadena positiva (+ssRNA) y su genoma tiene un tamaño de entre 27-32 kb. Desde el extremo terminal-5' vemos que dos tercios del genoma están formados por genes que codifican poliproteínas, pp1a y pp1b, que darán lugar a proteínas no estructurales necesarias para seguir el proceso de replicación viral. El extremo terminal-3' codifica las proteínas estructurales: proteína S o de espiga, proteína E o de envoltura, proteína M o de membrana y N o nucleocápside. Para poder codificar estas proteínas estructurales (y continuar el proceso de replicación) se necesitan genes accesorios que son específicos de cada especie. En la imagen de abajo aparecen diversas cepas de los géneros de coronavirus que conocemos. Como se puede apreciar en la imagen, las proteínas accesorias (3, 3a, 3b, 5, etc) pueden variar en cada especie.
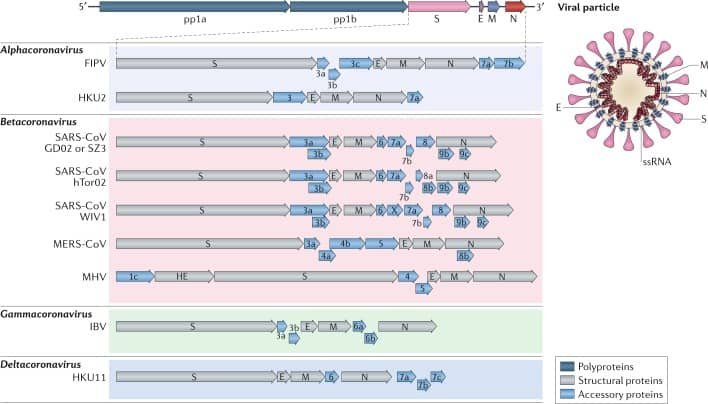

El ratio de mutaciones en los virus de ARN es muy superior a los de ADN; por ello, los genomas de virus ARN suelen ser muy cortos, de menos de 10 kb de largo. Sin embargo, los coronavirus tienen un genoma de unas 30 kb, lo que los hace los virus ARN más largos que se conozcan.
Ciclo de replicación viral
HCoV-NL63, SARS-CoV y SARS-CoV-2 se unen al receptor ACE-2 de los neumocitos tipo 2.
MERS-CoV se une al receptor DPP4 (dipeptidil peptidasa-4), una glicoproteína transmembrana de los neumocitos tipo 2.
HCoV-OC43 reconoce ciertos azúcares.
Respecto a los coronavirus animales, éstos se unen a otros receptores, como la aminopeptidasa N (TGEV, PEDV, y PRCV) o CEACAM1 (MHV). También pueden tener mismos receptores que los coronavirus humanos, como DDP4 (HKU4) o azúcares (BCoV).

(a) Clasificación de los coronavirus. Los coronavirus que aparecen de arriba a abajo son: NL63 (HCoV-NL63), coronavirus porcino de gastroenteritis transmisible (TGEV), virus de la diarrea epidémica porcina (PEDV), coronavirus respiratorio porcino (PRCV) del género Alphacoronavirus; coronavirus agudo severo relacionado con el síndrome respiratorio (SARS-CoV), Coronavirus causante del síndrome respiratorio de Oriente Medio (MERS-CoV), coronavirus de murciélago HKU4, virus de la hepatitis del ratón (MHV), coronavirus bovino (BCoV), coronavirus humano OC43 del género Betacoronavirus; coronavirus aviar de bronquitis infecciosa (IBV) del género Gammacoronavirus; deltacoronavirus porcino (PdCV) del género Deltacoronavirus.
(b) Estructura de la proteína S. Aparecen la subunidad de unión al receptor S1, la subunidad de fusión a la membrana S2, el ancla transmembrana (TM), la cola intracelular (IC), y la envuelta vírica.
(c) Esquema de los dominios estructurales de los picos de los coronavirus, incluyengo el dominio N-terminal S1 (S1-NTD), el dominio C-terminal S1 (S1-CTD), el péptido de fusión (FP), y las repeticiones heptad en las regiones N y C (HR-N and HR-C). Las tijeras indican dos sitios de proteólisis.
(d) Resumen de las estructuras y funciones de las proteínas S. Los receptores reconocidos por cada una de las subunidades S1 de arriba a abajo son: enzima convertidora de angiotensina 2 (ACE2), aminopeptidasa N (APN), dipeptidil peptidasa-4 (DPP4), molécula 1 de adhesión celular relacionada con el antígeno carcinoembrionario (CEACAM1), y azúcares. (Li F., 2016)
El ejemplo que hemos cogido es el de SARS-CoV-2, así que se muestran las proteínas accesorias y los receptores específicos de esta especie. Como hemos visto, cada coronavirus puede tener proteínas accesorias y receptores diferentes.
En el primer paso de unión al receptor, la proteína S se divide en S1 y S2 por proteasas celulares del huésped, como la serin-proteasa transmembrana 2 (TMPRSS2). S1 tiene como función principal la de unirse a los receptores de la superficie celular del hospedador, mientas que S2 se encarga de mediar la fusión membranal. SARS-CoV-2 parece tener gran afinidad por el receptor celular humano ACE-2 (enzima convertidora de angiotensina), y este es el lugar por donde entra. Los receptores ACE-2 aparecen en los neumocitos tipo 2 (células alveolares).
Las primeras proteínas que se traducen son las poliproteínas replicasas 1a (pp1a) y 1aab (pp1ab). Estas proteínas sufrirán un proceso de proteólisis que darán lugar a proteínas no estructurales, proteínas para continuar con la replicación como la ARN polimerasa dependiente de ARN, helicasas y proteínas no estructurales 3, 4 y 6 (nsP3, nsP4, nsP6). Se cree que nsP3, nsP4 y nsP6 anclan el complejo de replicación/ transcripción de SARS-CoV-2 por el reclutamiento de membrana del retículo endoplasmático para formar vesículas de doble membrana (DMV). La ARNpol y las helicasas están en los DMV, e impulsan la creación de ARN cuya traducción dará lugar a proteínas estructurales y accesorias.
Las proteínas S, E y M se insertan y se pliegan en el retículo endoplasmático, que luego se transportan al compartimento intermedio retículo endoplasmático-Golgi (ERGIC). Las proteínas N se unen al ARN genómico viral en el citoplasma y forman la nucleocápside. Más tarde, ocurre el ensamblaje final del virión en el ERGIC, y los viriones maduros se liberan por vesículas mediante exocitosis.

Infección: transmisión y síntomas
En general, los alfacoronavirus y betacoronavirus pueden infectar a mamíferos, mientras que los deltavirus y gamacoronavirus infectan aves, si bien algunos también pueden infectar mamíferos. En los humanos los alfacoronavirus y betacoronavirus pueden infectar los sistemas nervioso, respiratorio, gastrointestinal y hepático. Para llegar a infectar humanos los coronavirus deben recombinarse antes. Así, puede haber infecciones de animal a humano y entre humanos.
En el caso de los animales domésticos, la mayoría de coronavirus provocan enfermedades gastrointestinales. Esos son los casos del coronavirus porcino de gastroenteritis transmisible (TGEV), coronavirus bovino (BCoV), coronavirus felino (FCoV), coronavirus canino (CCoV) y el coronavirus del pavo (TCoV). No obstante, también pueden afectar a otras partes del cuerpo. Esos son los casos del virus de la bronquitis infecciosa (IBV) que causa bronquitis infecciosa en pollos, coronavirus respiratorio canino (CRCoV) que causa problemas respiratorios en perros, y el virus de la hepatitis de ratón (MHV) que puede causar encefalitis desmielinizante en ratones.
En humanos los coronavirus suelen afectar en especial al tracto respiratorio superior y al tracto gastrointestinal. Los síntomas pueden ser leves, como los de un resfriado común, hasta llegar a bronquitis, e incluso a neumonías con afectación renal. La transmisión principal es por las gotas respiratorias que expulsamos al hablar, toser o estornudar. De ahí la importancia de lavarse las manos a menudo, toser en el codo, y otras medidas de prevención son útiles contra estos virus.
HCoV
En este momento se conocen 4 coronavirus causantes del resfriado común: HCoV-229E (229E), HCoV-OC43 (OC43), HCoV-NL63 (NL63) y HCoV-HKU1 (HKU1). Estos virus no suelen ser muy peligrosos.
HCoV-229E. Está dentro del género Alphacoronavirus. Los síntomas de su infección incluyen malestar, dolor de cabeza, descarga nasal, estornudos y dolor de garganta. Algunos pacientes también desarrollan tos y fiebre. El tiempo de incubación oscila entre 2 y 5 días.
HCoV-OC43. Está dentro del género Betacoronavirus, en el lineaje A. Los síntomas son los mismos que 229E.
HCoV-NL63. Está dentro del género Alphacoronavirus. Este coronavirus está asociado principalmente con niños, ancianos y pacientes inmunocomprometidos con enfermedades respiratorias. Además de los síntomas de un resfriado se puede ver laringitis obstructiva.
HKU1. Está dentro del género Betacoronavirus, en el lineaje A. Los síntomas que se muestran con esta infección son leves. Los cuatro coronavirus están distribuidos a nivel mundial.
SARS-CoV
Provoca el síndrome agudo respiratorio severo (SARS). Está enfermedad respiratoria severa apareció en China en 2002. Después se propagó a otros países de Asia, además de América del Norte y Europa durante 9 meses. Los pacientes con esta infección presentaron fiebre, mialgia, dolor de cabeza y malestar general, seguido de tos seca, disnea y distrés respiratorio unos 5-7 días después que puede resultar en muerte. En algunos casos se incluyeron infecciones del tracto gastrointestinal, hígado, riñón y cerebro. En el pulmón se vio un aumento de macrófagos, proliferación epitelial y daño en la difusión alveolar. Su R0 es 3, aunque con las medidas de prevención llegó a ser inferior a 1.
La principal transmisión del SARS fue nosocomial, en hospitales. Parece ser que fue por el tratamiento a los pacientes sin las medidas de seguridad adecuadas, lo que explica que fueran frecuentes las infecciones del personal sanitario.
Se reportaron 8273 casos con 775 muertes; un ratio de mortalidad del 9%. La mayoría de casos ocurrieron en China y Hong Kong. Los ancianos fueron más susceptibles a esta infección, con un ratio de mortalidad del 50%.
MERS-CoV
Provoca el síndrome respiratorio de Oriente Medio (MERS). Está enfermedad respiratoria severa apareció en Oriente Medio en 2012. La primera vez que se observó fue en la muestra pulmonar de un paciente de 60 años que falleció por una enfermedad respiratoria grave en Jeddah, Arabia Saudí. Las manifestaciones clínicas de esta infección van desde pacientes asintomáticos hasta una neumonía severa con distrés respiratorio agudo, shock séptico y fallo renal que acaba en muerte. Los síntomas más frecuentes fueron fiebre, tos, dolor de garganta, dolor articular y muscular. Un tercio de los pacientes desarrolló síntomas gastrointestinales, como diarrea y vómitos. El fallo renal grave fue uno de los síntomas más llamativos de MERS-CoV, ya que no aparece en las infecciones humanas de coronavirus. Su R0 es de 0.7, inferior al del SARS y COVID-19.
La principal transmisión del MERS fue nosocomial, en hospitales, al igual que el SARS. En el caso de MERS hubo más contagios entre pacientes. Además, un análisis de las superficies de los hospitales mostró la presencia del virus en el ambiente días después de que los pacientes dejaran de dar positivo.
Hasta diciembre de 2015 se confirmaron 1621 casos confirmados en laboratorio, con 584 muertes, a lo largo de 26 países. El índice de mortalidad de este virus está sobre un 36%, lo que lo hace uno de los virus más peligrosos contra los humanos que conocemos.
SARS-CoV-2
Aquí tienes un artículo sobre la transmisión de SARS-CoV-2.
Tratamiento
Actualmente no existe ningún tratamiento contra ninguno de los coronavirus. En algunos no es tan urgente encontrarlo, en otros sí. En el caso de los coronavirus causantes del resfriado común, el tratamiento que se sigue es bastante sencillo: hidratación, reposo y algún antiinflamatorio si se llega a necesitar. En el caso de los otros coronavirus pueden hacer falta medicamentos, en especial en casos graves. Se han usado anticuerpos, interferones, inhibidores de proteasas virales y del huésped.
Aquí tienes un artículo sobre los tratamientos que se estudian para COVID-19. Algunos de los tratamientos contra la COVID-19 se usaron con anterioridad contra SARS y MERS.
En Arabia Saudí y China se usó ribavirina y algunos tipos de interferones (IFNs) contra MERS, en combinación con antibióticos de amplio espectro y oxígeno. La ribavirina también se uso con frecuencia cuando apareció el brote de SARS-CoV, en combinación con corticoesteroides que tenían efecto antiinflamatorio. El uso de IFNα se hacía en combinación con inmunoglobulinas o timosinas para estimular el desarrollo de células T, aunque algunas veces se hacía también con ribavirina. Ninguno de estos tratamientos ha tenido estudios clínicos así que es difícil asegurar su eficacia, y, además, los datos de los pacientes parecían tener ciertas contradicciones. En todo caso, la consideración principal es el uso de ribavirina siempre en combinación con otro tratamiento antiviral. Aunque los IFNs se han mostrado efectivos contra MERS-CoV in vitro (e incluso su efectividad aumenta junto a la ribavirina) no se ha probado su efectividad en humanos.
Otros antivirales han sido probados, como la combinación de ritonavir y lopinavir, inhibidores de la proteasa usados para tratar el VIH. En el caso del SARS, se mostró una mejora en los pacientes en los que se combinaban ritonavir y lopinavir con ribavirina, en comparación con ribavirina como único tratamiento. En Corea del Sur se usó una combinación de IFNα, ritonavir, lopinavir y ribavirina para tratar el MERS y parece que se recuperó un alto porcentaje de los pacientes con dicho tratamiento.
Otros medicamentos han mostrado inhibir la replicación de MERS-CoV in vitro, en especial la cloroquina, la clorpromazina, la loperamida y la ciclosporina A. No obstante sus mecanismos de acción no se conocen bien. La inhibición de TMPRSS2, una proteasa del hospedador aumenta la supervivencia en modelos murinos contra SARS. En este caso, se usa el mesilato de Camostat.
Bibliografía
Bukhari, K., Mulley, G., Gulyaeva, A. A., Zhao, L., Shu, G., Jiang, J., et al. Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. Virology 524, 160–171. 2018. doi: 10.1016/j.virol.2018.08.010
Chen Y, Liu Q, Guo D. Emerging coronaviruses: genome structure, replication, and pathogenesis. J Med Virol. 2020. https://doi.org/10.1002/jmv.25681
Cui, J., Li, F., & Shi, Z. L. (2019). Origin and evolution of pathogenic coronaviruses. Nature reviews. Microbiology, 17(3), 181–192. https://doi.org/10.1038/s41579-018-0118-9
de Wit, E., van Doremalen, N., Falzarano, D., & Munster, V. J. (2016). SARS and MERS: recent insights into emerging coronaviruses. Nature reviews. Microbiology, 14(8), 523–534. https://doi.org/10.1038/nrmicro.2016.81
Fung To S., Liu Ding X. Coronavirus infection, ER stress, apoptosis and innate immunity. Frontiers in Microbiology. 2020. DOI: https://doi.org/10.3389/fmicb.2014.00296
Habibzadeh P, Stoneman EK. The novel coronavirus: A bird's eye view. Int J Occup Environ Med 2020;11:65-71. 2020. doi: 10.15171/ijoem.2020.1921
Li F. (2016). Structure, Function, and Evolution of Coronavirus Spike Proteins. Annual review of virology, 3(1), 237–261. https://doi.org/10.1146/annurev-virology-110615-042301
Su S, Wong G, Shi W, Liu J, Lai ACK, Zhou J, Liu W, Bi Y, Gao GF. Epidemiology, genetic recombination, and pathogenesis of coronaviruses. Trends Microbiol. (2016) ;24(6):490–502. DOI:https://doi.org/10.1016/j.tim.2016.03.003
